发布日期:2025-04-22 浏览次数: 信息来源:科技成果部
近日,中国农业大学理学院数学与交叉科学研究中心、应用数学系王涵副教授在统计基因组学和生物信息学方向顶级期刊《基因组生物学》(Genome Biology)上发表了题研究论文《MAAT:一种在全转录组关联性分析中整合多种功能注释信息的非参数贝叶斯方法》(MAAT: a new nonparametric Bayesian framework for incorporating multiple functional annotations in transcriptome-wide association studies)的研究论文。
该研究提出了一种名为MAAT的非参数贝叶斯方法,通过整合多种基因组功能注释信息显著提升了全转录组关联性分析(TWAS)的功效,并通过一种基于角度的度量方式解析了基因影响性状时起关键作用的注释信息,为解析疾病的遗传机制提供了新工具。MAAT突破了现有的TWAS方法假设注释信息与SNP影响效应呈线性关系的局限性,为在TWAS分析中整合多种注释数据提供了新的方案。
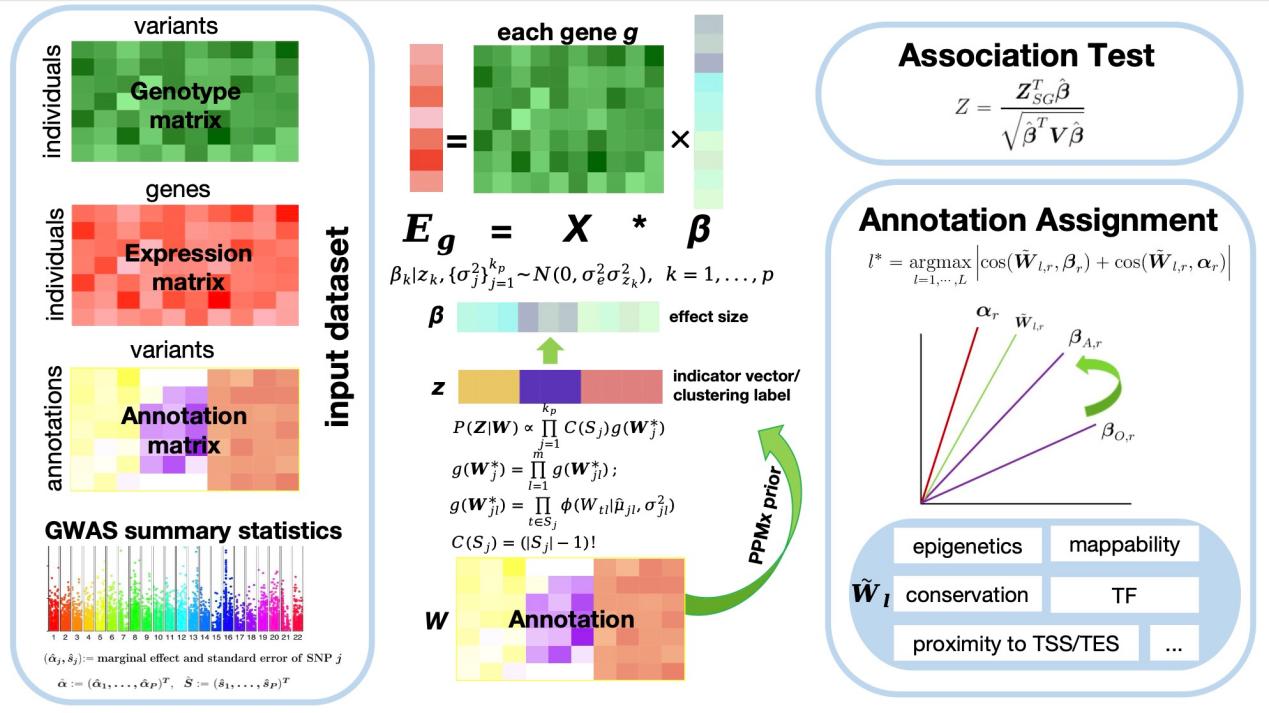
MAAT流程图
在后GWAS时代,TWAS将基因表达作为中介物,是连接GWAS发现的大量的非编码区变异与复杂疾病的关键桥梁。现有的添加基因组注释信息的TWAS方法大多依赖表观遗传注释,并假设注释信息与SNP效应大小呈线性关联。MAAT通过引入PPMx非参数贝叶斯先验,可以将表观遗传信息、进化保守性、转录因子结合等七类功能注释整合到TWAS框架中,根据注释相似性对SNP进行聚类,使注释特征相近的SNP共享对基因表达的效应值。摆脱了线性假设束缚,可灵活捕捉复杂的注释-效应关系。
将MAAT应用到八类脑疾病中(如阿兹海默症、精神分裂症等),发现了许多新的TWAS位点,ARL13B, SPI1, LRRC37A, NSF等基因可以在多种脑疾病中发挥作用。MAAT也发现了在许多已被证实的遗传相关性很强的疾病中,TWAS显著基因有大量的重叠,如双向情感障碍和精神分裂症,神经性厌食症和抑郁症。研究团队也为每个基因-疾病的显著关联赋予了一个最重要的注释信息,这为研究疾病的机理起到了一定的参考作用。
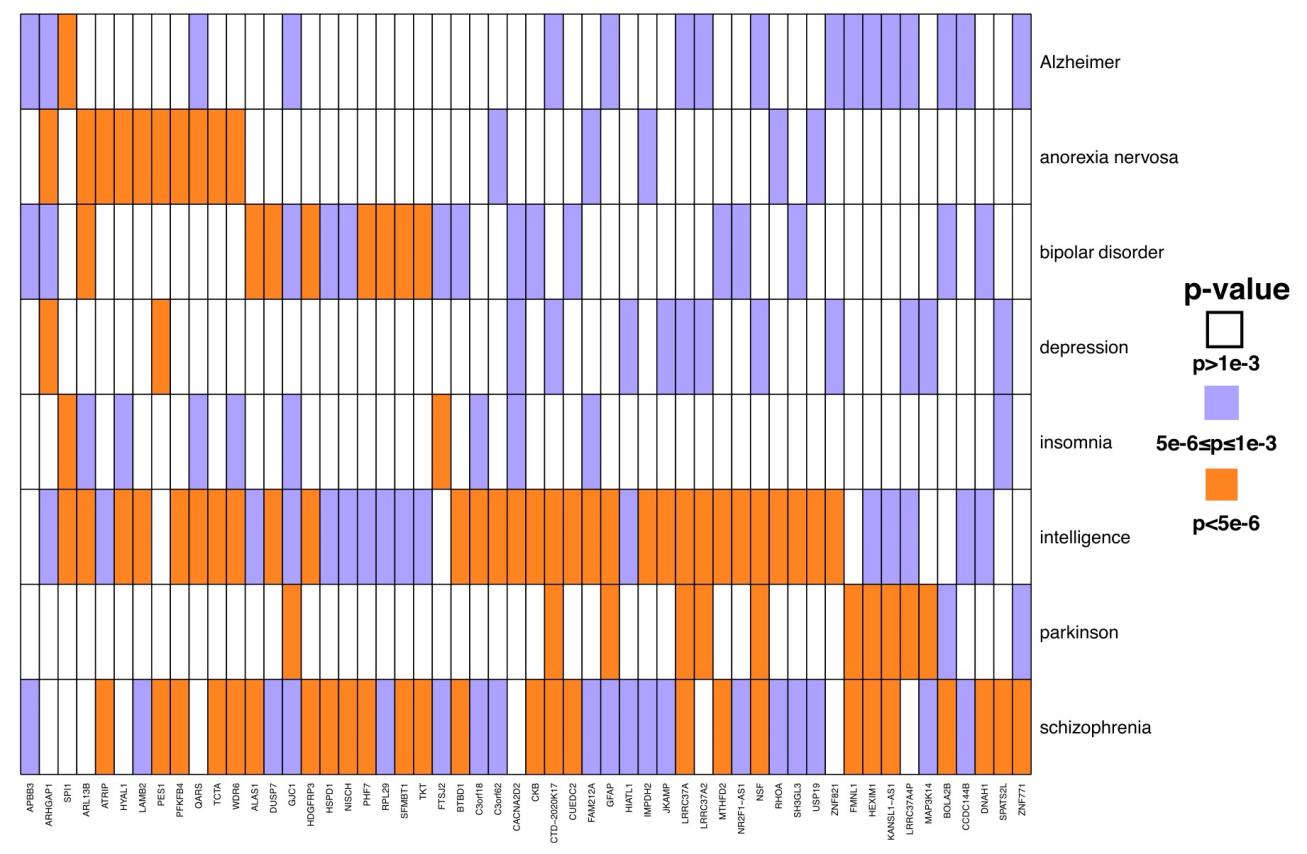
MAAT找到的在多种脑疾病中均显著相关的基因
MAAT展望了三大方向:结合单细胞转录组数据进行TWAS分析、进行跨种族扩展验证,以及开发TWAS精细定位工具进行下游处理。MAAT提出的非参数贝叶斯框架也将为生物数据的整合提供新的工具。
中国农业大学为论文第一完成单位,中国农业大学理学院数学与交叉科学研究中心、应用数学系王涵副教授为论文第一作者,香港大学计算与数据科学学院统计系张彦教授为论文通讯作者,香港大学医学院Pak Chung Sham教授、香港大学统计系李响博士、北京协和医学院李腾博士和李哲博士为论文做出了重要贡献。
数学与交叉科学研究中心2024年10月成立以来,积极推进跨学科研究合作,已与农学院、生物学院、食品学院等多个学院的研究团队建立了合作关系。目前,相关交叉研究课题正在有序推进中。为进一步深化跨学科研究,中心计划在本学期拓展合作范围,与更多学院开展学术交流与合作,共同推动交叉科学研究的创新发展。